专家笔谈 | 中国新生儿基因组计划:罕见病基因型与表型队列研究的新进展
罕见病联盟
罕见病作为全球公共卫生关注的重点,对儿童群体尤其是新生儿群体产生了巨大影响。近年来迅速发展的二代测序技术为罕见病的早期发现和精准治疗提供了更可行更全面的手段。当前,世界各地开展了诸多新生儿基因组计划,旨在探索基因测序技术在遗传性罕见病诊断中的价值和优势。在中国,中国遗传学会遗传咨询分会联合复旦大学附属儿科医院推出了10万例目标的中国新生儿基因组计划(CNGP)。该计划综合持续收集的大规模基因组数据和详细表型信息,对新生儿罕见病进行深入的基因型-表型相关性研究,为早期揭示罕见病遗传病因和实现精准治疗提供依据,同时也为药物基因组学、成人发病疾病提供预测。本文将基于CNGP已发表的多项新生儿罕见病基因型-表型队列研究,对基因型-表型采集流程和既往队列研究进展进行概述,并对当前新生儿罕见病研究的不足进行总结,从而为今后研究方向提供参考。
罕见病是全球卫生公共事业的关注热点,全世界共有近3亿罕见病患者[1-2]。50%的罕见病在出生或儿童时期发病,且病情进展迅速,病死率高,30%的患儿寿命不超过5岁[3]。针对罕见病的早诊早治,除了应用新生儿筛查(newborn screening,NBS)在新生儿出生后对某些危害严重的先天性遗传疾病进行群体检测之外,近年来迅速发展的二代测序(next-generation sequencing,NGS)技术凭借其准确、全面的变异检测特点,在罕见病精准诊治及遗传疾病防控窗口前移方面展现出明显优势。
新生儿是儿科罕见病研究的一个重要群体,当前世界各地针对新生儿群体的基因组项目正在陆续进行,主要研究目的包括:①提高新生儿重症监护室(neonatal intensive care units,NICU)患者孟德尔疾病的诊断率,以及评估使用NGS加速诊断流程的益处[4-5];②使用基因组测序估计“表观健康”,即表面上看起来健康的、无明显健康问题或症状的婴儿的未来健康风险[6];③统一纳入健康和不健康的新生儿进行兼具诊断和预测目的的研究[7-8]。涉及的疾病包括新生儿免疫缺陷[9]、先天性代谢缺陷[6,10]、先天性耳聋[6,11]、儿童癌症易感综合征[2]和新生儿肥厚型心肌病[12]等。
迄今国际上开展的超10万例样本的大规模新生儿基因组计划包括美国的GUARDIAN研究(https://guardian-study.org)、英国的新生儿基因组计划(Newborn Genomes Programme,https://www.genomicsengland.co.uk/initiatives/newborns),以及中国遗传学会遗传咨询分会联合复旦大学附属儿科医院发起的中国新生儿基因组计划(China Neonatal Genomes Project,CNGP)[13]。CNGP期望在NGS技术成熟的基础上,建立中国特有的新生儿基因组数据库,搭建新生儿遗传病基因检测工作流程,推动新生儿遗传病基因检测产业化,完善遗传咨询培训体系[13-14]。
本文将基于CNGP已发表的多项新生儿罕见病基因型-表型队列研究,对基因型-表型采集流程和既往队列研究进展进行概述,并对当前新生儿罕见病研究的不足进行总结,从而为今后研究方向提供参考。
1新生儿罕见病队列研究中的基因型-表型采集策略
1.1基因型采集策略
推动精准医学的落实需要实现基因型与表型的精确采集与对应。目前,主要的基因型采集策略包括临床外显子组测序(clinical exome sequencing,CES)[15-16]和基因包检测[17]、全外显子组测序(whole exome sequencing,WES),以及全基因组测序(whole genome sequencing,WGS)等。
CES和基因包检测都选择性地测序特定基因区域,主要用于某类特定的或已知的与疾病密切相关的基因检测。当前CES已能实现同时检测单核苷酸变异(single nucleotide variant,SNV)和拷贝数变异(copy-number variant,CNV),具有时间短、成本低的特点[18]。目前CES的遗传诊断率为20%~40%[18-21]。经先证者CES检测到的可疑病因通过补充家系CES,即同时对父母和子女进行CES,能进一步提高诊断率[19]。
WES对基因组编码区进行检测,涵盖超过20 000个基因,比CES检测范围更广,可确定25%~40%患者疾病的遗传基础[22]。人类疾病全外显子组约占全基因组的2%,但85%左右的基因突变却发生于外显子区,因此对于编码基因的变异研究,采用WES是经济高效的选择。对于异质性的疾病,或者多系统受累的复杂疾病,尤为适合使用WES,更能帮助研究者挖掘和探索罕见病新基因[23]。
WGS是目前检测范围最广的分子基因检测技术,可同时对编码区和非编码区DNA进行测序,非编码区对于调控基因表达和维持基因组稳定有着不可或缺的作用[24]。但因全基因组变异信息量庞大,传统WGS检测需要投入大量的人力与时间,花费较高昂。而快速全基因组测序(rapid whole genome sequencing,rWGS)在WGS的基础上加速数据处理和分析,当前最快诊断时间可缩短至20 h[25]。rWGS的合理应用可实现临床紧急情况下的快速决策[26],减少不必要的经验性治疗,并加快遗传和预后咨询的进程[4,27]。
基于CNGP建立的高通量测序流程自2015年推出第1版,并经过两次更新升级之后,已累计实现临床应用超10万例[28-31]。CES主要用于病情较轻的普通新生患儿,针对重症人群则使用覆盖范围更广的WES和WGS[13]。
1.2表型采集策略
新生儿罕见病队列的表型采集是一项复杂的任务,涉及临床文书的记录、实验室检验检查结果的汇总、影像学资料的分析、多组学数据的集成,以及患者随访信息的追踪。具体来说,临床文书包括病史、临床表现和治疗反应;检验检查包括一系列疾病生化、免疫学特征及分子生物学标记有关的血液、尿液等生物样本分析;影像信息包括X射线、CT、核磁共振;多组学分析应用高通量技术研究转录组(基因表达)、蛋白质组(蛋白质表达和修饰)和代谢组(代谢物变化)等,提供关于生物体在分子水平上的综合信息;随访信息包括定期的临床评估、实验室检查和影像学检查的更新,以及患者生活质量和生存状态的记录。
CNGP采集的新生儿表型主要包含3类疾病,具体如下:①发病率高的疾病,如遗传代谢疾病、免疫缺陷疾病、智力发育障碍等;②在中国人群中出现的特殊疾病,如地中海贫血、葡萄糖-6-磷酸脱氢酶缺乏症等;③目前未知的疾病。整个工作流程包括建立研究的纳排标准、从患者电子病历中提取人类表型本体(human phenotype ontology,HPO)术语,最后集成到自身开发的PhenoPro表型评分算法以形成完整的表型收集链[32]。具体来说,CNGP的纳入标准包括:①新生儿(年龄≤28 d);②中国父母;③获得至少2 mL静脉血样本;④父母或监护人知情同意。排除标准:①多胎妊娠的母亲;②18岁以下无法做出同意决定的父母;③拒绝将遗传数据用于后续研究分析的患者或监护人[13]。通过临床病历中的表型为每个新生儿自动分配HPO术语,对指定HPO术语可能影响的疾病进行优先级排序,使用疾病-基因关系推断候选致病基因的P值,结合变异过滤程序选择的罕见且可能有害的候选致病变异的P值,从而进行孟德尔疾病的基因诊断[32]。
CNGP纳入的新生儿主要具有以下表型:①颅面畸形;②中枢神经系统异常:脑发育不良、脑积水、惊厥/癫痫、肌张力低下/肌张力过高、颅内出血、中枢神经系统感染等;③心血管异常:先天性心脏病、心肌病、心律失常、心功能不全等;④代谢性疾病:串联质谱分析怀疑先天性代谢异常、高血糖/低血糖、严重电解质紊乱、代谢性酸中毒、高氨血症和甲状腺功能障碍;⑤消化系统异常:消化系统畸形、腹裂/疝气、喂养问题、肝脾肿大、胆汁淤积、反复腹泻等;⑥呼吸系统异常:后鼻孔闭锁、喉软化、肺发育不全、气胸、胸腔积液、先天性中枢性低通气综合征、严重呼吸衰竭;⑦骨骼异常:骨骼发育不良、关节畸形、指(趾)畸形、骨折等;⑧泌尿生殖系统异常:肾发育不良、输尿管异常、尿道下裂、隐睾、肾功能不全等;⑨感染和免疫受累:细胞或体液免疫缺陷、败血症;⑩血液学异常:凝血异常、贫血/红细胞增多症、血小板减少症、弥散性血管内凝血、噬血细胞综合征等[33]。
随着对表型研究的不断深入,近年来已涌现许多利用人工智能和机器学习的技术,实现精细化表型提取的研究,例如,与遗传综合征相关的面部特征研究[34],以及与肢体生长基因相关的指纹图案研究[35]。更加完整的表型信息有助于开发基因优先级排序算法,而对临床表型进行分类和标准化则能够更有效地促进临床信息的系统记录和分析[36]。
2基于中国新生儿基因组计划的中国新生儿遗传罕见病基因型-表型队列研究进展
CNGP的实行涉及两个阶段,第一阶段为2016年8月至2021年12月,对招募的30 000名疑似遗传病的新生儿进行CES或WES检测;第二阶段计划于2022年1月至2025年12月进行,使用CES对70 000名新生儿进行遗传病筛查。目前,10万例测序目标已提前完成。
2.1基于CNGP的NICU队列
在新生儿疾病研究方面,借力于自主开发的新生儿高通量分子诊断平台、全国共享的中国儿童罕见病数据库等成果,已有多篇新生儿队列研究发表,涵盖国际最大规模的NICU队列、全国最大样本量最大覆盖面的极早产儿队列,以及新生儿神经发育障碍队列、新生儿死亡队列、小于胎龄儿队列等(表1)。CNGP的发展为精准化诊疗策略提供了许多根据,包括靶向药物治疗、饮食调整、造血干细胞移植,以及启动多学科团队随访等多种医疗干预措施[33]。具体案例包括为ALDH7A1基因致病变异使用大剂量维生素B6,针对KCNQ2基因致病变异使用卡马西平控制癫痫发作,对导致庞贝病的GAA基因致病变异、导致Shwachman-Diamond综合征的SBDS基因致病变异进行酶替代治疗[50],以及对IL2RG基因致病变异导致的X连锁严重联合免疫缺陷病改常规抗生素治疗为抗结核和干细胞移植治疗,对OTC基因致病变异导致的尿素循环障碍进行肝移植治疗,对遗传预后不良的婴儿转向姑息治疗等[54]。
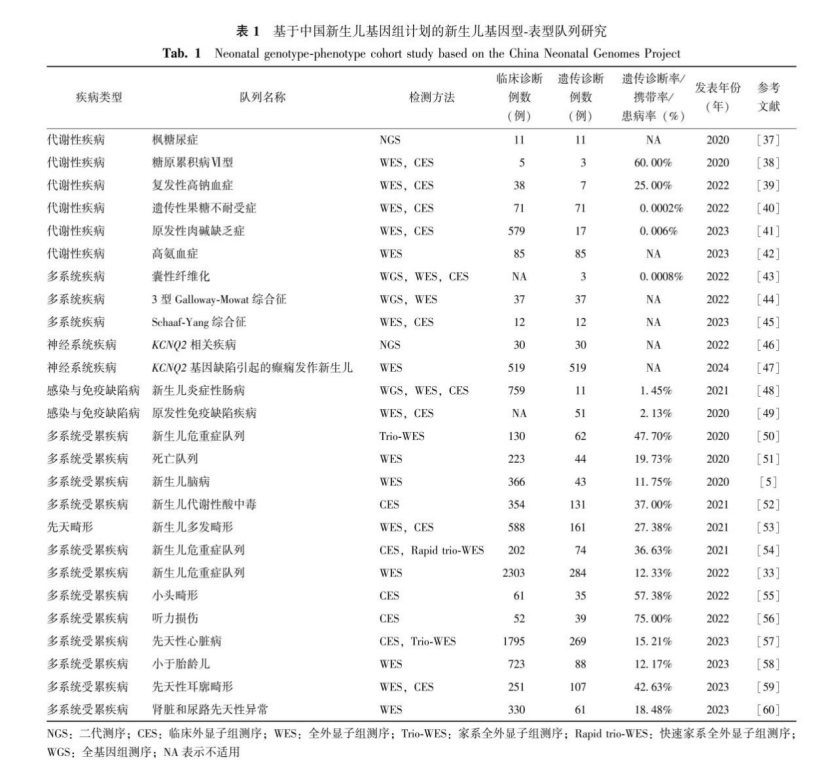
表 1基于中国新生儿基因组计划的新生儿基因型-表型队列研究
Tab.1 Neonatal genotype-phenotype cohort study based on the China Neonatal Genomes Project
2.2基于CNGP的罕见病队列
在罕见病队列方面,基于CNGP的罕见病队列研究已有多篇报道,涉及疾病包括遗传性果糖不耐受症、原发性肉碱缺乏症、囊性纤维化、枫糖尿症、糖原累积病Ⅳ型,以及Schaaf-Yang综合征等。此外,还有多项研究结合了CNGP数据库中本土人群数据和公共数据库的国际报告数据,估计罕见病在中国人群中的患病率,并进行基因型-表型的相关性分析。
例如,Tang等[40]通过整合 3 例来自CNGP和68例来自文献发表的患者遗传信息,并通过华表计划、 gnomAD数据库获取中国人群携带频率,计算出中国遗传性果糖不耐受症患病率约为1/504 678,并通过基因型-表型分析发现携带ALDOB基因c.448G>C(p.A150P)纯合致病变异位点的患者更容易出现恶心,携带两个错义致病变异位点的患者更容易出现对甜食和水果的厌恶。Ji等[41]通过整合来自CNGP数据库和人类基因突变数据库(human gene mutation database,HGMD)、ClinVar、Web of Science和PubMed的278个原发性肉碱缺乏症相关的SLC22A5基因变异,借助自主开发的变异评级流程鉴定出128个致病或可疑致病变异。该研究发现中国人群的原发性肉碱缺乏症患病率为1/17 456,远高于白种人群,同时揭示中国人群中SLC22A5的热点变异为c.1400C>G (p.S467C)、c.51C>G(p.F17L)和c.760C>T(p.R254*),也与白种人群的报道存在差异。Ni等[43]通过分析CNGP的30 951份WES样本,利用CFTR基因的致病或可疑致病变异位点估算出中国人群的囊性纤维化患病率为1/128 434,并确定外显子区域rs213950(c.1408G>A,p.V470M)和rs1042077(c.2562T>G,p.T854=)为疾病相关的单核苷酸多态性位点(single nucleotide polymorphism,SNP)。
在罕见病致病变异挖掘方面,涉及对11名枫糖尿症[37]、5名糖原累积病Ⅳ型[38]、12名Schaaf-Yang综合征[45]进行回顾性的基因型-表型探索,分别发现了8个枫糖尿症相关的BCKDHA、BCKDHB和DBT基因的新变异位点,一个新的糖原累积病Ⅳ型PYGL基因3.6 kb缺失热点变异,6种Schaaf-Yang综合征致病基因MAGEL2新致病变异。这些探索使4名枫糖尿症胎儿得到了产前诊断,证实3.6 kb缺失与糖原累积病Ⅳ型的关联并提高疾病的分子诊断率,分析出中国Schaaf-Yang综合征新生儿的典型特征和主要死亡原因。此外,基于罕见病致病基因的研究还有助于新生儿预后管理。KCNQ2基因与良性新生儿癫痫发作及癫痫性脑病有着紧密关联。在基于CNGP的NICU遗传缺陷研究中,KCNQ2被鉴定为致病变异频率最高的基因[33]。Huang等[47]基于KCNQ2基因缺陷构建癫痫新生儿神经发育预后模型,发现蛋白质的功能和结构特性,以及变异的致病性,相比单纯的临床信息预测具有更高的预测准确性,这一模型为新生儿癫痫发作后的神经发育预后管理提供了新的参考。
3新生儿罕见病基因型-表型队列研究的不足和展望
目前,基因组检测还只能对30%~50%疑似罕见遗传病的患者做出诊断[1],这表明还有50%以上的患者无法获得精确的遗传学解释和相应的医疗指导。当前诸多研究结果固然能促进人们对罕见病遗传背景的认识,也极大地推动了罕见病的早筛、早诊和早治,但罕见病药物基因组学、复杂疾病分析、晚发性遗传病等各类问题还有待解决。
罕见病研究的不足一方面表现为表型数据采集相对单一,无法全面捕捉疾病的多维度特征。涵盖儿童从出生到成长关键时刻的体格发育、运动发育的实验室检查和影像学完整数据尚未完善,收集生化检验、血常规、脑电数据、行为学测量等指标的远程方法尚未建立。另外,能够识别与特定遗传综合征相关的高精度人脸识别技术有待开发,这些技术的应用有助于增强表型数据的维度和质量,提高某些特定疾病的诊断率。另一方面,新生儿基因组测序项目尚未形成长期的随访计划。目前的随访计划仅通过访谈收集有限的表型信息,如家庭生活、发育状况等,导致后续随访的信息管理费时费力,影响研究结果的可比性和可靠性,更可能使一些患者无法得到明确有效的诊断。
未来,需要将普通疾病队列、高危疾病队列与遗传队列进行有机衔接,在出生时即对生物标志物和预警指标进行检测以便及时干预,建立高危患儿的纳排标准。同时形成一套可在基层推广的基因检测方法,结合质谱数据、蛋白组学数据和脑电图信息等,在日常医疗工作的基础上,对队列进行纵向跟踪。完整的新生儿队列可以反哺产前队列,未来将队列研究真正向全人群铺开、推广,还需要更多的思考和更多的设计,也需要更多的机构共同推动。
作者贡献:董欣然负责完成文章撰写;周文浩负责审核。
利益冲突:所有作者均声明不存在利益冲突。
参考文献
[1]Stark Z,Scott RH. Genomic newborn screening for rare diseases[J]. Nat Rev Genet,2023,24(11):755-766.
[2]Yeh JM,Stout NK,Chaudhry A,et al. Universal newborn genetic screening for pediatric cancer predisposition syndromes:model-based insights[J]. Genet Med,2021,23(7):1366-1371.
[3]Elliott AM. Genetic counseling and genome sequencing in pediatric rare disease[J]. Cold Spring Harb Perspect Med,2020,10(3):a036632.
[4]Petrikin JE,Cakici JA,Clark MM,et al. The NSIGHT1-randomized controlled trial:rapid whole-genome sequencing for accelerated etiologic diagnosis in critically ill infants[J]. NPJ Genom Med,2018,3:6.
[5]Yang L,Chen X,Liu X,et al. Clinical features and underlying genetic causes in neonatal encephalopathy:a large cohort study[J].Clin Genet,2020,98(4):365-373.
[6]Roman TS,Crowley SB,Roche MI,et al. Genomic sequencing for newborn screening: results of the NC NEXUS project[J]. Am J Hum Genet,2020,107(4):596-611.
[7]Mei H,Dong X,Wu B,et al. Clinical and genetic etiologies of neonatal unconjugated hyperbilirubinemia in the China Neonatal Genomes Project[J]. J Pediatr,2022,243:53-60.e9.
[8]Ceyhan-Birsoy O,Machini K,Lebo MS,et al. A curated gene list for reporting results of newborn genomic sequencing[J]. Genet Med,2017,19(7):809-818.
[9]Pavey AR,Bodian DL,Vilboux T,et al. Utilization of genomic sequencing for population screening of immunodeficiencies in the newborn[J]. Genet Med,2017,19(12):1367-1375.
[10]Adhikari AN,Gallagher RC,Wang Y,et al. The role of exome sequencing in newborn screening for inborn errors of metabolism[J]. Nat Med,2020,26(9):1392-1397.
[11]Downie L,Halliday J,Lewis S,et al. Exome sequencing in newborns with congenital deafness as a model for genomic newborn screening: the Baby Beyond Hearing Project[J]. Genet Med,2020,22(5):937-944.
[12]Christensen KD,McMahon PM,Galbraith LN,et al. Benefits,harms,and costs of newborn genetic screening for hypertrophic cardiomyopathy:estimates from the PreEMPT model[J]. Genet Med,2023,25(4):100797.
[13]Xiao F,Yan K,Wang H,et al. Protocol of the China Neonatal Genomes Project:an observational study about genetic testing on 100,000 neonates[J]. Pediatr Med,2021,4:28.
[14]Dong X,Xiao T,Chen B,et al. Precision medicine via the integration of phenotype-genotype information in neonatal genome project[J]. Fundamental Research,2022,2(6):873-884.
[15]O'Donnell-Luria AH,Miller DT. A clinician's perspective on clinical exome sequencing[J]. Hum Genet,2016,135(6):643-654.
[16]Timmermans S. Trust in standards:transitioning clinical exome sequencing from bench to bedside[J]. Soc Stud Sci,2015,45(1):77-99.
[17]Saudi Mendeliome Group. Comprehensive gene panels provide advantages over clinical exome sequencing for Mendelian diseases[J]. Genome Biol,2015,16(1):134.
[18]Dong X,Liu B,Yang L,et al. Clinical exome sequencing as the first-tier test for diagnosing developmental disorders covering both CNV and SNV: a Chinese cohort[J]. J Med Genet, 2020, 57(8):558-566.
[19]Lee H,Deignan JL,Dorrani N,et al. Clinical exome sequencing for genetic identification of rare Mendelian disorders[J]. JAMA,2014,312(18):1880-1887.
[20]Rudilla F,Franco-Jarava C,Martínez-Gallo M,et al. Expanding the clinical and genetic spectra of primary immunodeficiency-related disorders with clinical exome sequencing:expected and unexpected findings[J]. Front Immunol,2019,10:2325.
[21]Mahfouz NA,Kizhakkedath P,Ibrahim A,et al. Utility of clinical exome sequencing in a complex Emirati pediatric cohort[J]. Comput Struct Biotechnol J,2020,18:1020-1027.
[22]Sawyer SL,Hartley T,Dyment DA,et al. Utility of whole-exome sequencing for those near the end of the diagnostic odyssey:time to address gaps in care[J]. Clin Genet,2016,89(3): 275-284.
[23]Chang HR,Cho SY,Lee JH,et al. Hypomorphic mutations in TONSL cause SPONASTRIME dysplasia[J]. Am J Hum Genet,2019,104(3):439-453.
[24]Hartley T,Lemire G,Kernohan KD,et al. New diagnostic approaches for undiagnosed rare genetic diseases[J]. Annu Rev Genomics Hum Genet,2020,21:351-372.
[25]Clark MM,Hildreth A,Batalov S,et al. Diagnosis of genetic diseases in seriously ill children by rapid whole-genome sequencing and automated phenotyping and interpretation[J]. Sci Transl Med,2019,11(489):eaat6177.
[26]Farnaes L,Hildreth A,Sweeney NM,et al. Rapid whole-genome sequencing decreases infant morbidity and cost of hospitalization[J]. NPJ Genom Med,2018,3:10.
[27]Saunders CJ,Miller NA,Soden SE,et al. Rapid whole-genome sequencing for genetic disease diagnosis in neonatal intensive care units[J]. Sci Transl Med, 2012, 4(154): 154ra135.
[28]黎籽秀,刘博,徐凌丽,等.高通量测序数据分析和临床诊断流程的解读[J].中国循证儿科杂志,2015,10(1):19-24.
[29]黎籽秀,刘博,杨琳,等.高通量测序数据分析和临床诊断流程对新生儿多发畸形候选变异的筛选准确性研究[J].中国循证儿科杂志,2015,10(1):25-28.
[30]杨琳,董欣然,彭小敏,等.复旦大学附属儿科医院高通量测序数据分析流程(第二版)对遗传疾病候选变异基因筛选用时和准确性分析[J].中国循证儿科杂志,2018,13(2):118-123.
[31]陈宾,董欣然,王慧君,等.复旦大学附属儿科医院高通量测序数据一体化全流程闭环分析系统及临床应用案例分析[J].中国循证儿科杂志,2022,17(3):202-209.
[32]Li Z,Zhang F,Wang Y,et al. PhenoPro:a novel toolkit for assisting in the diagnosis of Mendelian disease[J]. Bioinformatics,2019,35(19):3559-3566.
[33]Yang L,Wei Z,Chen X,et al. Use of medical exome sequencing for identification of underlying genetic defects in NICU:experience in a cohort of 2303 neonates in China[J]. Clin Genet,2022,101(1):101-109.
[34]Gurovich Y,Hanani Y,Bar O, et al. Identifying facial phenotypes of genetic disorders using deep learning[J].Nat Med,2019,25(1):60-64.
[35]Li J,Glover JD,Zhang H,et al. Limb development genes underlie variation in human fingerprint patterns[J]. Cell,2022,185(1):95-112.e18.
[36]Haendel MA,Chute CG,Robinson PN. Classification, ontology, and precision medicine[J]. N Engl J Med,2018,379(15):1452-1462.
[37]Sun WH,Wu BB,Wang YQ,et al. Identification of eight novel mutations in 11 Chinese patients with maple syrup urine disease[J]. World J Pediatr,2020,16(4):401-410.
[38]Liu B,Wu B,Lu Y,et al. A novel, recurrent, 3.6-kb deletion in the PYGL gene contributes to glycogen storage disease type Ⅵ[J]. J Mol Diagn,2020,22(12):1373-1382.
[39]Hu L,Yang L,Yan K,et al. Importance of early genetic sequencing in neonates admitted to NICU with recurrent hypernatremia:results of a prospective cohort study[J]. Neonatology, 2022, 119(1):103-110.
[40]Tang M,Chen X,Ni Q,et al. Estimation of hereditary fructose intolerance prevalence in the Chinese population[J]. Orphanet J Rare Dis,2022,17(1):326.
[41]Ji X,Ge Y,Ni Q, et al. Primary carnitine deficiency:estimation of prevalence in Chinese population and insights into newborn screening[J]. Front Genet,2023,14:1304458.
[42]Li M,Chen X,Chen H,et al. Genetic background and clinical characteristics of infantile hyperammonemia[J]. Transl Pediatr,2023,12(5):882-889.
[43]Ni Q,Chen X,Zhang P,et al. Systematic estimation of cystic fibrosis prevalence in Chinese and genetic spectrum comparison to Caucasians[J]. Orphanet J Rare Dis,2022,17(1):129.
[44]Xu S,Hu L,Yang L,et al. Galloway-Mowat syndrome type 3 caused by OSGEP gene variants:a case report and literature review[J]. Front Pediatr,2022,10:899991.
[45]Huang Z,Lu W,Zhang P,et al. Early onset critically ill infants with Schaaf-Yang syndrome: a retrospective study from the China neonatal genomes project and literature review[J]. Ann Transl Med,2023,11(9):312.
[46]Xiao T,Chen X,Xu Y,et al. Clinical study of 30 novel KCNQ2 variants/deletions in KCNQ2-related disorders[J]. Front Mol Neurosci,2022,15:809810.
[47]Huang Z,Liu B,Xiao T,et al. Neurodevelopmental outcomes prediction in newborns with seizures caused by KCNQ2 gene defects[J]. Neonatology,2024,121(2):178-186.
[48]Peng X,Lu Y,Wu B,et al. A novel 333 bp deletion of IL10RA in Chinese patients with neonatal-onset inflammatory bowel disease[J]. J Clin Immunol,2021,41(5):1095-1098.
[49]Sun J,Yang L,Lu Y,et al. Screening for primary immunodeficiency diseases by next-generation sequencing in early life[J]. Clin Transl Immunology,2020,9(5):e1138.
[50]Wang H,Lu Y,Dong X,et al. Optimized trio genome sequencing (OTGS) as a first-tier genetic test in critically ill infants:practice in China[J]. Hum Genet,2020,139(4):473-482.
[51]Yang L,Liu X,Li Z,et al. Genetic aetiology of early infant deaths in a neonatal intensive care unit[J]. J Med Genet,2020,57(3):169-177.
[52]Ma H,Tang Z,Xiao F,et al. Neonatal metabolic acidosis in the neonatal intensive care unit: what are the genetic causes?[J]. Front Pediatr,2021,9:727301.
[53]Wang H,Xiao F,Dong X,et al. Diagnostic and clinical utility of next-generation sequencing in children born with multiple congenital anomalies in the China Neonatal Genomes Project[J]. Hum Mutat,2021,42(4):434-444.
[54]Wu B,Kang W,Wang Y,et al. Application of full-spectrum rapid clinical genome sequencing improves diagnostic rate and clinical outcomes in critically Ill infants in the China Neonatal Genomes Project[J]. Crit Care Med,2021,49(10):1674-1683.
[55]Ye C,Mei H,Chen H,et al. Molecular genetic analysis of newborns with congenital microcephaly[J]. Neonatology,2022,119(4):455-463.
[56]Zhu Y,Hu L,Yang L,et al. Association between expanded genomic sequencing combined with hearing screening and detection of hearing loss among newborns in a neonatal intensive care unit[J]. JAMA Netw Open,2022,5(7):e2220986.
[57]Wang H,Xiao F,Qian Y,et al. Genetic architecture in neonatal intensive care unit patients with congenital heart defects: a retrospective study from the China Neonatal Genomes Project[J]. J Med Genet,2023,60(3):247-253.
[58]Xiao H,Chen H,Chen X,et al. Comprehensive assessment of the genetic characteristics of small for gestational age newborns in NICU: from diagnosis of genetic disorders to prediction of prognosis[J]. Genome Med,2023,15(1):112.
[59]Xiao F,Wu B,Dong C,et al. Genetic spectrums and clinical profiles of critically ill neonates with congenital auricular deformity in the China Neonatal Genomes Project[J]. Hum Genet, 2023,142(12):1737-1745.
[60]Huang Z,Shen Q,Wu B,et al. Genetic spectrum of congenital anomalies of the kidney and urinary tract in Chinese Newborn Genome Project[J]. Kidney Int Rep,2023,8(11): 2376-2384.


